磁気ビーズによるDNA精製
「エタチン」と言えば分子生物学を実習レベルであれ経験したことのある全ての日本人が知る専門用語だろう。エタノール沈殿の略語である。塩とエタノールによるDNA析出作用を用いて、溶液中のDNAを選択的に取り出す手法で、常温で15分くらいでやったり、-80˚Cで1時間置いたり、ラボごとにさまざまな流儀がある。
一方、DNAシークエンシングの現場ではエタノール沈殿ではなくPEG(ポリエチレングリコール)沈殿を使うことが多い。それも、SPRI (Solid-phase reversible immobilization: 固相可逆固定化)ビーズという磁気ビーズを用いる方法が一般的だ。これは
- エタノール沈殿よりも微量での回収効率が高い(実際私もpgレベルの超微量DNAで高収率で得られることを経験している)
- 遠心機を使わず、8連ピペットを用いた多検体同時処理に向いている
- 用いるPEG・ビーズ溶液の量によって、簡単にDNAのサイズ選択が行える
点が大きい。とりわけ、3のサイズ選択ができる点はプライマーやプライマーダイマーを除きながら目的産物だけを抽出したり、テンプレートのゲノムDNAを除いたりなどが精製のステップでできるのが大きい。元のDNA溶液に対して1.8Xのビーズ溶液を使うと100bp以上のDNAがほぼ100%、0.8Xだと200bp以上が、0.6Xだと350bp以上が、そして下限値の0.45X(この量を扱う時には超厳密に液量調整が必要)の場合には500bp以上が回収できるというわけだ。なので、私のラボではとりあえず学生さんも新人の技術員さんも、SPRIビーズ精製でDNA回収率が85%以上になるまで練習していただくところから始めるくらいの基礎技術になっている。SPRIビーズ精製もTipsはたくさんあるが、
- 溶液は全て常温にしてしっかり混ぜる
- 80%エタノールは都度調整する
- ビーズペレットを乾かしすぎない(エタノールは飛ばすが水は飛ばさない、程度)
- Elutionは長いDNAの場合には37˚Cで15分
- (別件だが)DNAシークエンス関連試薬はEDTAが干渉する反応が多いのでElution BufferにEDTAは使わない
あたりが基本的なコツになる。
このSPRIビーズ精製、基本的にBeckman Coulter社のAgencourt AMPure XPという商品と同義である。DNAシークエンシングと言えばAMPure XP、といわんばかりに、デファクトのキットになっている。ラボでもSPRIビーズ精製と言わずに「AMPure精製」と普段から言ってしまうくらいに。ところがどっこい、このAMPure XP、大変お高い。5mLで5万円強、450mLだと100万円くらいする。最も高価なワインの一つとして有名なDRC ロマネ・コンティが750mlで300万円くらいのご時世なので、5mL単位でAMPure XPを買うと余裕でmL単価はロマネ・コンティが安価になるくらい。50µl 反応系のPCR後精製を1X AMPure XPでやると5mLで100回できるので一回あたり500円。こうやってみても結構高い。
私の研究室ではしかし、もうエタ沈をすることはほとんどなく、普段のPCR後精製を含め常にSPRIビーズ精製をしている。もちろんこれは私たちが大富豪なわけではなく、SPRIビーズ溶液を自作しているのである。SPRIビーズ溶液の自作プロトコルはOpenWetWareなどにもあるので比較的良く知られているかと思うが、参考までに以下にうちで使っているプロトコルを。
- GE Healthcare Sera-Mag SpeedBead Carboxylate-Modified Magnetic Particles (Hydrophobic) 15ml を購入
- Sera-Magビーズ1mLを10mM Tris-HClでマグネットスタンドを使って3回ウォッシュ(ビーズは毎回マグネットスタンドから外し良くピペッティングして混ぜる・上清は毎回捨てる)
- 50mLチューブに25mL 5M NaClを入れる
- 洗って乾いたSera-Magビーズに1ml 10mM Tris-HCl pH 8.0を入れ、スタンドから外し良く混ぜ、全量をStep3の50mLチューブに加える
- さらにオートクレーブ済み20mL 50% (w/v) PEG 8000 in 10mM Tris-HCl pH 8.0を加える。粘性が高いので注意。
- 4mL 10mM Tris-HCl pH 8.0を入れ、最終液量を50mLにする
以上を電気泳動用ラダーを使い、1.8X, 1X, 0.8X, 0.6Xで精製(サイズセレクション)し、目的サイズがちゃんと選択されていることを確認してから使用。使用までは冷蔵庫で保存。これだと750mL分が約8万円で作れるので、AMPure XPの10~100倍安い。ちなみに、AMPure XPにはさらに高いRNA用のRNAClean XPという兄弟品があるが、これはRNA分解活性がないことが検査されているだけで中身は同じなので、サイズ確認と同時にRNA分解検査をやっておくとなおベター。
さて、ここまではすでに実践済みの方も多いかと思うので追加でもう一つ。Illuminaシークエンシングの際には100~1000bpあたりのレンジのサイズセレクションができれば十分であったが、ロングリードが当たり前の昨今、10kbpなどの長いDNAは残し、短いDNAを捨てたいというケースが多々ある。AMPure XPは基本的に上述の通り、磁気ビーズとPEG(20% PEG8000)と塩(2.5M NaCl)であり、加える量でDNAのサイズが選択されているのは基本的に塩の作用である(0.45Xが下限なのはおそらく析出に必要なPEGの下限量)。なので、上述のPEGと塩の量を変えることで、より広範囲なサイズセレクションが可能になる。
実はこの辺も既に緻密な研究が報告されているので、興味がある方はこちらの論文:
SPRI Beads-based Size Selection in the Range of 2-10kb – PMC (nih.gov)
のFig. 2をご覧あれ。ただ、塩梅は結構シビアなので必ず電気泳動様ラダーなどで確認することを忘れずに。15kbp以下のDNAを捨てるようなキットも販売されているが、原理がわかってしまえば容易にラボにあるもので代替できるし、現状商品化されていない1~10kbpの間のサイズセレクションも自在に行える。以下、あるプロジェクトで必要になって4kbp以上のサイズセレクションを実際に私のラボでやった例を最後にご紹介。
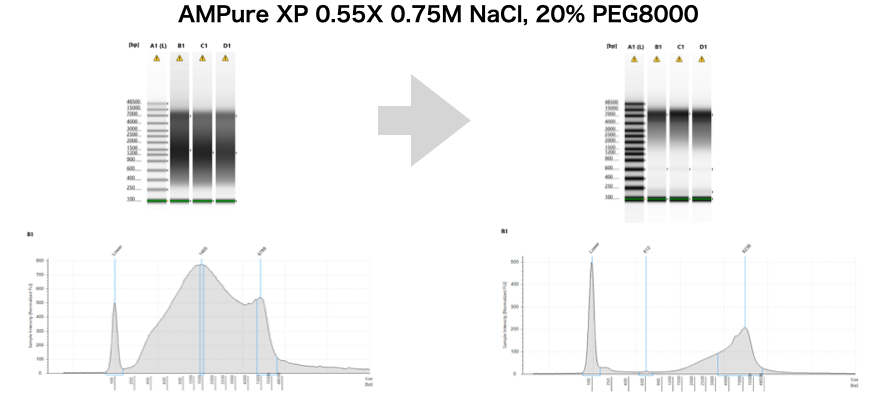
投稿者プロフィール