iGONAD2022 (1) IDT homepageを使ったゲノム編集のデザインとBenchlingの使い方
東海大の大塚さんと鹿児島大の佐藤さんの芸術作品とも言える簡易ゲノム編集動物作製メソッドのiGONAD。当ラボに導入してからはや3年が経ちましたが、その間にもこれすごい、マジかよ、うわっ、私でもできた!という声が巷で続々。この再現性の良さと手軽さと革新性は、今から20年ちょい前に東北大の仲村春和さんが広めたニワトリ胚へのエレクトロポレーションによるプラスミド導入法を彷彿させます。iGONADの熱烈なファンの一人として、2022年度版ゲノム編集による変異マウスの作製法、覚え書きも含めてこちらにまとめておきたいと思います。
まずゲノム編集のデザインから。今のところうちのラボでうまくいっているのは
– insert-deletionによる変異
– タグ配列やストップコドンの挿入変異
– 点変異の導入
– poly-A配列やU6でドライブしたsmall RNAなど~300ntまでのカセットの挿入
などですが、鋳型となるssODNさえ準備できれば、2 kbぐらいまでの挿入も可能なようです。ここではうちで一番実績のあるタグ配列の挿入を例にとって説明します。
具体的にどのようにデザインしていくかですが、まず遺伝子配列をとってくるところから。これは無料で利用できるクラウドサービス、Benchlingが超オススメです。以前はEnsemblのページからエクソンイントロン情報をちまちまコピペで移していたのですが、データベースから一発importできます。有料のプラスミド管理ソフトでもできるのでしょうが、あまりの優秀な機能と操作性に、うちではこちらに完全移行。Gene Construction Kitなどのgccファイルもimportできるので、移行はスムーズです。具体例があったほうがわかりやすいと思いますので、lncRNAだと思っていたら機能未知の新規ペプチドだったよーんのChd3OS、またの名をnf227のC末端にFLAGタグ配列を挿入するデザインを作ってみます。
まずはBenchlingのimport DNA sequenceの機能を使ってゲノム情報をとってきます。かんたん、かんたん。
次に、編集したい部位からだいたい前後100塩基ぐらいとってきて、合計200塩基をIDTのAlt-R™ HDR Design Toolにぶっこみます。そうするとその付近の配列が画面に出てきますので、挿入部位を正確に合わせ、ノックインしたい配列を入力します。FLAGタグなどはすでに配列があるのでプルダウンメニューでオーケーです。左右のアームの長さですが、HDR Design Toolのデフォルトだと40ntで設計するようですが、ssODNは200ntまで注文できるので、なるべく長く、75ntにしています。null変異体を作りたいときは、開始メチオニンの数塩基下流にこれでもかこれでもかとストップコドンを入れた5′-TAGcTGAcTAAgctTGAcTAGcTAA-3’配列をmutationのボックスにカスタムで入力してます。ちなみにHindIIIサイトが有るのは制限酵素サイトがないと安心できない世代の職業病です。ちなみにAlt-R™ HDR Design Toolのページに飛ぶためにはIDTへのアカウント登録とログインが必要なようです。そうすると、、、
こんな感じで、適切なcrRNAの配列と相同組換えのためのssODNの配列を出してくれます。なんともeasyで拍子抜けなぐらいです。このツールが出る前は、crRNAを同じくIDTのCustom Alt-R® CRISPR-Cas9 guide RNAで選択したあとで、挿入部位の前後のアームの配列の領域を決め、挿入部位がcrRNAにかかるならそのまま、かからないのであれば相同組換後の配列の再切断を防ぐためにPAM配列に変異を入れたり、組換後のcrRNAの配列にアミノ酸置換を伴わないサイレント変異を入れたり、、、といろいろ頭を悩ます、でも楽しい作業があったのですが、素人の猿知恵よりはこちらのほうが適切な配列を返してくれるんでしょう。きっと。有用なツールは効率的ではありますが、しばしば実験のささやかな楽しみを奪ってしまうものです。ああ古の100本ミニプレップ、、、
追記:デフォルトのセッティングだと、crRNAの切断効率やオフターゲットよりも挿入部位の近さを優先するみたいで、トップで出てくるデザインはウームという気がしなくもない、です。そういう場合は、この記事の一番下の方にあるAlt-R Custom Cas9 crRNA Design ToolでcrRNAを選んで、そのcrRNAをgRNAの配列で指定してやるとよいかと思います。
あとは、IDTが返してきたssODN (Alt-R™ HDR Donor Oligos)とcrRNAを注文するだけ。オーダーはポチポチボダンを押すだけです。ここで、(+)鎖をたのむか、(-)鎖を頼むかが重要で、crRNAの向きに関わらず、Cas9の切断部位よりも5’側にノックインしたいときは、(+)鎖を、3’側にノックインしたいときは(-)鎖の配列をオーダーする必要があります。理由は簡単で、相同組換が起きるときは切断部位から5′ to 3’のEXO nucleaseが働いて修復に必要なプライマーの3’末端が出てくるので、(+)鎖がプライマーとなる場合は切断部位よりも3’側に、(-)鎖がプライマーとなる場合は切断部位よりも5’側が修復されていきます。ssODNはプライマーと相補的な配列になりますので、(+)ssODN for 5′ Kin, (-)ssODN for 3′ Kinとなるわけです。
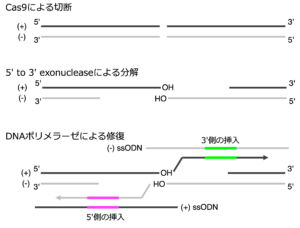
ここでちょっと悩ましいのが、crRNAとssODNの関係です。上述の通り、ssODNのstrandはcrRNAのstrandに依存せず、切断部位と挿入部位の関係によって強制的に決まってしまうので、crRNAとssODNが相補的な配列を持つ場合と、同じ鎖の配列を持っている場合の両方が考えられます。相補的な配列を保つ場合は、Cas9に取り込まれたcrRNAがssODNと複合体を作るという文献的な報告もあり、ssODNが効率的に核に運ばれてるので効率が良くなるようです。ただ、直感的には、その場合ssODNが標的のdsDNAとの結合を競合的に阻害するような気もします。あまりそのへんは気にしていなかったのですが、例数も溜まってきたので、ノックイン配列がPCRで確認できた個体の割合をちょっとまとめてみました。横軸は切断部位と挿入部位がどれぐらい離れているかの距離、縦軸はF0個体でノックイン配列が見られた割合、sはcrRNAとssODNが同じstrand、asは相補鎖だった場合です。

うーん。どうなんでしょう。ノックイン効率は切断部位から離れると少しずつ悪くなるようですが、strandはあんまり大きな差はないような気がします。crRNAの配列も違いますし、システマチックに調べたわけではないので、ほんの参考程度にしかならないかもしれませんが、そもそもiGONADの効率がものすごく良いので、多少効率に差はあるのかもしれないけれども2-3腹ゲノム編集すれば目的のものは取れるので結果オーライ、といったところでしょうか。
次に、IDTにオーダーした配列の情報をBenchlingにも移しておきます。まず、ssODNの配列をコピペして新しいBenchlingファイルを作ります。次に、Benchlingのalignmentの機能を使うと元のファイルとのアラインメントを返してくれるので、armの配列を確認して、Benchlingのannotation機能を使って領域にannotationをつけていきます。挿入部位の配列が挿入配列と同じ場合、アラインメントがずれてることがあるので、そのあたりマニュアルで補正する必要があります。
さて、最後にゲノム編集チェック用のPCRのプライマーのデザインです。デザインはNCBI primer BLAST (https://www.ncbi.nlm.nih.gov/tools/primer-blast/)が便利です。左右両方についてarmの外側の配列と挿入配列を増幅できるプライマーを選択します。left arm側の挿入をチェックする場合、left armの100 bp上流からright armまでの250 bpをコピーして、NCBIのprimer BLASTに投げると便利です。forwardのプライマーを1-100 bp(armの外側)からピックアップ、reverseのプライマーを175 bpから250 bp(挿入配列を挟んで反対側のarm)からピックアップ、というような感じで検索することができます。同様に、right arm側の挿入をチェックす場合は、left armからright armの100塩基下流の250 bpの配列をコピーして、forwardを1-75 bpで、reverseを150-250 bpで検索すればオーケーです。ん、このデザインだとarmの長さが53 bpと78 bpになってますね。ホントのことをいうとちょっとずらさないといけない、、、けれども、まあ、大体の領域はあっているので、目的のプライマーはピックアップされているようです。genotyping時に短い断片のほうが効率よく増幅されるので、長さは200 bpをMaxにしてます。今回はマウスなので、ゲノムにmm10を指定しています。結果が出てきたらBenchlingにアノテーションをつけておきます。プライマーが見つかりませんでしたよ、と言われたら、Tmの下限値と上限値の幅を動かして、FWとRVのTmの差も緩めると、なんとか見つけてきてくれます。
あとはIDTから届いたオリゴを溶かして、duplex作って、Cas9と混ぜてcomplex作って、、、ですが、このあたりは次回。
補足:crRNAだけデザインしたいという需要もあるかと思いますが、その場合もIDTのAlt-R Custom Cas9 crRNA Design Tool がとても便利です。Benchlingでゲノム編集したい領域近辺100 bpぐらいとってきて、mouseゲノムを選択して、クリックするだけ。off targetリスクとon target効率の両方の基準を満たす、下の動画でいうと白色のボックスのところのcrRNAを選べばオーケーです。昔はcrRNA、というかgRNAを選んだらT7アッセイで効率を確かめてから次のステップに進んでいましたが、今はIDTのこのツールでpick upされたcrRNAを選んで外れだったことはないので、T7アッセイは省略してます。時々、白いボックスのところに一つもcrRNA候補がない、あってもゲノム編集部位からかなり離れている、というときもありますが、その場合は、赤い領域でも境界領域、ひとマス目のところにあるやつを選んでも、うまくいっています。ちなみにオーダーすると、何故かデフォルトでは2 nmolを3本注文するようになっていますが、いいんですか、ほんとに3本でなくていいんですか、という声を無視して1本で十分です。また、他にもこんなのありますよ、いいんですか、ほんとに買わなくていいんですか、というリストが出てきますが、これも、ええ、結構です、とcheck outに進んで構いません。見てるといろいろ試したくなるんですけどね。セールスが上手いIDTさん。
投稿者プロフィール

最新の投稿
 ノンドメインブログ2025.06.27来し方行末~パート1
ノンドメインブログ2025.06.27来し方行末~パート1 ノンドメインブログ2025.02.11平和な日々
ノンドメインブログ2025.02.11平和な日々 ノンドメインブログ2024.11.25MBSJ2024のシンポジウムのすごいゲスト
ノンドメインブログ2024.11.25MBSJ2024のシンポジウムのすごいゲスト ノンドメインブログ2024.09.17奇遇癖
ノンドメインブログ2024.09.17奇遇癖


