目で数えることで見える世界
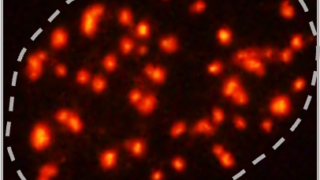
私たちは核内のDNA損傷とその修復について主に研究していますが、一つの問題は核という小さいスペースの中の、さらにDNA損傷が起こって反応が起こる範囲というのが非常に小さい範囲で、最近のイメージングの技術を用いてもまだまだ細かい部分の構造がよくわかっていません。DNA損傷のマーカーとしてγH2AXというものが昔からよく知られています。これはヒストンH2AのバリアントであるH2AXにリン酸化が入ったフォームを指しますが、このリン酸化はDNA二重鎖切断が入ったゲノム部分の両側に数百Kbpから数Mbpにわたって分布することが知られており、免疫染色をすると綺麗なフォーカスとして見えます(図1)。色々な研究から、このフォーカスは1対1でDNA二重鎖切断と対応していると考えられています。つまり、フォーカスの数は、その時点で存在するDNA二重鎖切断の数に相当するわけです。

γH2AXフォーカス
(点線は核を示す)
「フォーカスの数=未修復のDNA二重鎖切断数」ですので、野生型の細胞だと、例えば放射線照射によってDNA二重鎖切断を誘導した後には、順調にフォーカスの数が減少していく様子、つまり修復のキネティクスが解析できます。ところが、DNA二重鎖切断修復に異常があると、フォーカスの数が減少せず残るようになってしまい、修復がうまくいかず遅延していると解釈することができます。
DNA二重鎖切断修復の研究を始めたときに、まず習得したのは、放射線で誘導したDNA二重鎖切断に対応するγH2AXのフォーカスの数を顕微鏡下で数えるということでした。形も大きさも多様なフォーカスを数えるのは最初は非常に難しく、これを数えていいのか、これは数えなくていいのかと迷うことが多々ありました。しかもこの作業はかなりの忍耐が必要で、暗室にこもって細胞を何個も何個も何時間も数えていると、しまいには目をつぶってもフォーカスが瞼の裏に見えるように・・・。それに加えて、スライドの準備は比較的簡単な免疫染色で、色々なバックグラウンドの細胞(修復因子のノックアウト、ノックダウンなど)、DNA二重鎖切断誘導からのタイムポイントなど、多種多様な条件で実験をいくらでも組めてしまい、少しカウントをサボると、数えるべきスライドがあっという間に大量に溜まってしまいます。
同じ放射線量でDNA損傷を与えても、細胞によってγH2AXの数はかなりバラツキがあります。例えば平均20個と言っても、少ない細胞では12-3個かと思うと、30個超えるものもあります。ただそのバラツキは細胞に潜在的な多様性からきているようで、どんな条件で実験してもバラツキ具合は全く変わりません(なので平均の数で比較ができます)。カウントを続けていくとだんだんと慣れてきて、一細胞内にあるフォーカスの数が、数える前に大体何個くらいと分かるようになってきます。そして達人の域に達すると、野生型の細胞と比べて10-15%の増加(例えば野生型でγH2AXフォーカスが平均20個のところが22-23個)があるサンプルであれば、バラツキにもかかわらず、γH2AXで染色したスライドを遠目にパッと見た瞬間に修復が遅延していることがわかってしまいます(その結果、次の条件でスライドを作ってしまい、さらに宿題が・・・)。
今では画像解析の技術が発達してきたので、フォーカスの写真を撮って、後でまとめて画像処理にかけることで大まかな傾向はわかりますし、恣意的ともとれる顕微鏡下での目でのカウントを検証するという意味でも、画像によるカウントは重要なのは間違いありません。しかし、いくら大量の画像を処理して、統計処理して得られた有意差ありの10%の違いだけでは確信はなかなかもてず、一方、目で見た10%は、数えるまでもなく明らかという経験を何度もしてきました。
この目で数えて見つけた10%のカウントの違いにこだわって研究を続けた結果、新たなDNA二重鎖切断修復機構を2つ見つけることができたことは、単純、かつ時代遅れに見える作業も、案外侮れないということを実感する出来事となりました。
P.S. 先月より京都大学大学院生命科学研究科に異動しました。今後ともどうぞよろしくお願いいたします。
https://www.lif.kyoto-u.ac.jp/j/research/lab/6628/
http://www.rbc.kyoto-u.ac.jp/genome_stress/(近日中公開予定)