イネ免疫受容体の進化解析の論文を発表しました
植物は動物とは異なる戦略で免疫を発達させてきた。植物が持つ特徴的な性質として、受容体数が非常に多い点が挙げられる。細胞内免疫受容体であるNLRは、ヒトでは20遺伝子しか存在しないが、イネでは約500遺伝子も存在する。この遺伝子数の増加が植物免疫の頑健性に貢献していると推測されているが、増加したNLR遺伝子がどのような過程を経て新しい機能を獲得し、植物免疫に貢献しているかは十分に理解されていない。
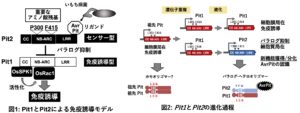
最近、我々らは、穀物のイネの重要病害であるいもち病菌に対する抵抗性遺伝子Pit1の隣に遺伝子重複で生まれ、進化の過程で機能が変化したパラログPit2遺伝子が存在することを発見した(Li et al., Nat Commn 2024)。興味深いことに、Pit1とPit2は直接結合し、ペアNLRタンパク質として機能していた。様々な解析から、Pit1が免疫誘導型であること、Pit2が病原体由来のリガンドを認識するセンサー型として機能することを発見した(図1)。Pit1とPit2のドメインスワップ実験の結果から、Pit1とPit2間の免疫誘導活性の有無を規定する運命決定残基を同定し、Pit2の免疫誘導活性の消失は、NB-ARCドメインに存在するP300とF415の2つのアミノ酸変異に起因し、この変異によりPit1とPit2が「パラログ抑制」の関係になったと推測された(図2)。Pit2の機能を規定する2残基の機能変化や進化の過程を解析することにより、祖先NLR遺伝子から遺伝子重複後に、免疫誘導型とセンサー型NLRが生まれた進化過程を明らかにできると考え、進化解析を行った。栽培イネや野生イネのゲノム配列情報を用いたPit1とPit2の進化解析により、アミノ酸が変化する非同義置換がPit2のLRRドメインに顕著に蓄積することを見出した。LRRドメインは、病原体由来のリガンドを認識する部位であることから、植物は遺伝子重複によりセンサー型NLR数を増加させることで、新たな病原体を認識することが可能になったと推測される。このように、遺伝子重複により生まれた遺伝子が新たな機能を持つことは、「新機能獲得(neofunctionalization)」と呼ばれる(図2)。平衡選択により多くのPit2の多型が集団内で維持できれば、より広範ないもち病菌を認識することが可能となり、イネの生存戦略としては非常に有利である。本研究は、遺伝子重複によるNLR遺伝子数の増加が植物免疫の頑健性に貢献するメカニズムを直接的に説明しうる事例となった。
論文タイトル:An NLR paralog Pit2 generated from tandem duplication of Pit1 fine-tunes Pit1 localization and function
雑誌:Nature Communications 2024 15(1):4610
論文リンク:https://www.nature.com/articles/s41467-024-48943-5
投稿者プロフィール
最新の投稿
 ノンドメインブログ2025.08.21相棒は生成AI? ある生物学研究者のサバイバル術
ノンドメインブログ2025.08.21相棒は生成AI? ある生物学研究者のサバイバル術 ノンドメインブログ2024.06.26イネ免疫受容体の進化解析の論文を発表しました
ノンドメインブログ2024.06.26イネ免疫受容体の進化解析の論文を発表しました


