先入観の罠
「来し方行く末」というとても深遠なテーマをいただき、これはChatGPTだろうと思ったのですがすでに取り上げられておりました・・・。そこで、未来のためには今をしっかり見る(先入観にとらわれずにデータを見る)ことが重要と思いますので、これにまつわる最近の気づきについて少しご紹介したいと思います。
我々の研究室ではPICを使ったRNA-seqを日常的にやっており、その経験からRINが5に達しないサンプルの解析は困難だと考えていました。その日も、とある先生から送っていただいた凍結切片のRINを測定したところ、RINは4前後と残念ながら分解の進んだサンプルであることが判明した折でした。このまま実験を進めてもまともなデータが出ないと危惧し、実験を続行するかどうか先方に掛け合ったところ「Visiumでは一応リードが取れるようなサンプルだった」とのこと。最終的にこれが決め手となりPIC RNA-seqを決行することとなりました。
出てきたシーケンス結果を見たところ、当初の不安は的中し、ほとんどのリードは遺伝子のエクソン部分にアサインされていません。その値はおよそ5%前後。心の中で「またつまらぬものを斬ってしまった・・・」と呟きました。どういうことかと言いますと、我々の解析パイプラインでは得られたシーケンスリードをリファレンスゲノムにマッピングする手法を採用しており、配列がRNAに由来するならばリードはゲノムのエクソン部分に集中してアサインされるはずなのです。逆に言えば、ゲノムDNAに由来する配列をマッピングさせても5%程度はエクソン部分にアサインされてしまいます。これはゲノムのおよそ5%がエクソン部分に相当するためです。つまり、「またつまらぬものを・・・」と私が思ったのはシーケンスした配列がRNA由来なのかDNA由来なのかよくわからない、解釈不能なデータの雰囲気を感じたためです。
そうは言っても結果は結果。先方とディスカッションする段取りを整え、詳細は当日ご説明しますということで解析データを先立って共有しました。そして迎えたディスカッション当日。沖さん(現所属の教授)と共にお通夜に参列するような面持ちで結果の説明を始めました。お叱りを受けるか、あるいは失望されるかと思いながら話を進めていくと、思いのほか結果に納得されている様子。実は先方でもデータを解析してくださっており、複数の遺伝子についてin situ hybridizationの結果と一致しているため今回の結果は悪くないのではないかと逆に元気付けていただきました。
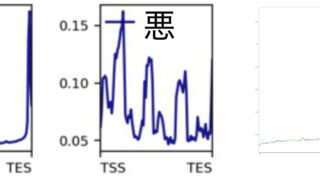
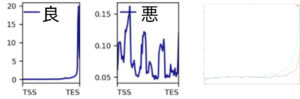
これまでの経験からアサイン率が悪ければ結果も悪いというのが我々の中で常識化していましたので、この朗報にはかなり衝撃を受けました。そこで何が起きているのか詳しく調べてみようということになりました。その結果わかったのは、エクソン部分にアサインされたリードはゲノムDNA由来ではなくちゃんとRNA由来だったということでした。これはエクソン部分にアサインされたリードが遺伝子のどこにマッピングされているかを解析することでわかりました(いわゆるカバレッジ解析)。PIC RNA-seqではRNAはpoly A 尾部から逆転写されるため、リードがRNAに由来する場合はその分布が遺伝子の終わり(3′末端付近)に集中します(図、左)。リードがゲノムDNAに由来する場合はこのような偏りはなく、遺伝子全域にわたってリードが分布します(実はゲノムのAリッチな配列付近に多くなる)(図、真ん中)。この方法で件のシーケンスデータを再解析したところ、アサインされたリードは遺伝子の終わり付近に集中していることがわかり(図、右)、珠玉の瓦礫にあるが如く5%は確かに意味のあるデータであることが確認できました。
今回のケースから先入観にとらわれずに生データを精査することの大切さを改めて痛感させられました。一方で、外部からの指摘がなければこのような気づきには至らなかったとも思われ、10年後の未来であっても個人の興味関心から発される問いかけによって初めて自覚させられる問題点についてはChatGPTは教えてくれないのではないかと思っています。