UTR の威力
大阪大学の加藤です。私たちはミジンコにおける遺伝子機能抑制実験、遺伝子機能獲得実験法を開発し、環境依存的な性決定メカニズムの研究を進めております。研究開始当初は不勉強で遺伝子発現制御といえばプロモーター領域、エンハンサー領域を介した転写制御を中心に考えておりましたが、遺伝子機能獲得実験法の開発の過程で遺伝子発現における非翻訳領域(UTR)の威力を痛感したちょっと古いエピソードがありますので、紹介をさせていただきたいと思います。
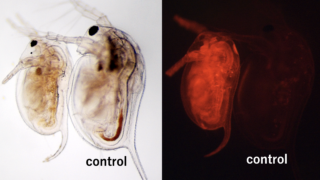
なんとかミジンコ卵へのマイクロインジェクションに成功し、次は長年楽しみにしていた蛍光タンパク質でミジンコを光らせることに挑みました。ミジンコの EST 解析で最も豊富であった EF1alpha1 mRNA の 5′ UTR と 3′ UTR を付加した DsRed2 のキメラ RNA を SP6 RNA polymerase により合成し、卵に注入するとインジェクションから 3 日後までもビカビカに光り、大興奮!ただし、1 つ気になることがありました。インジェクションした SP6 の転写産物、ゲル電気泳動ではバンドが 2 つ(完全長と同じサイズの full RNA、完全長より短いサイズの short RNA)見られたのです。
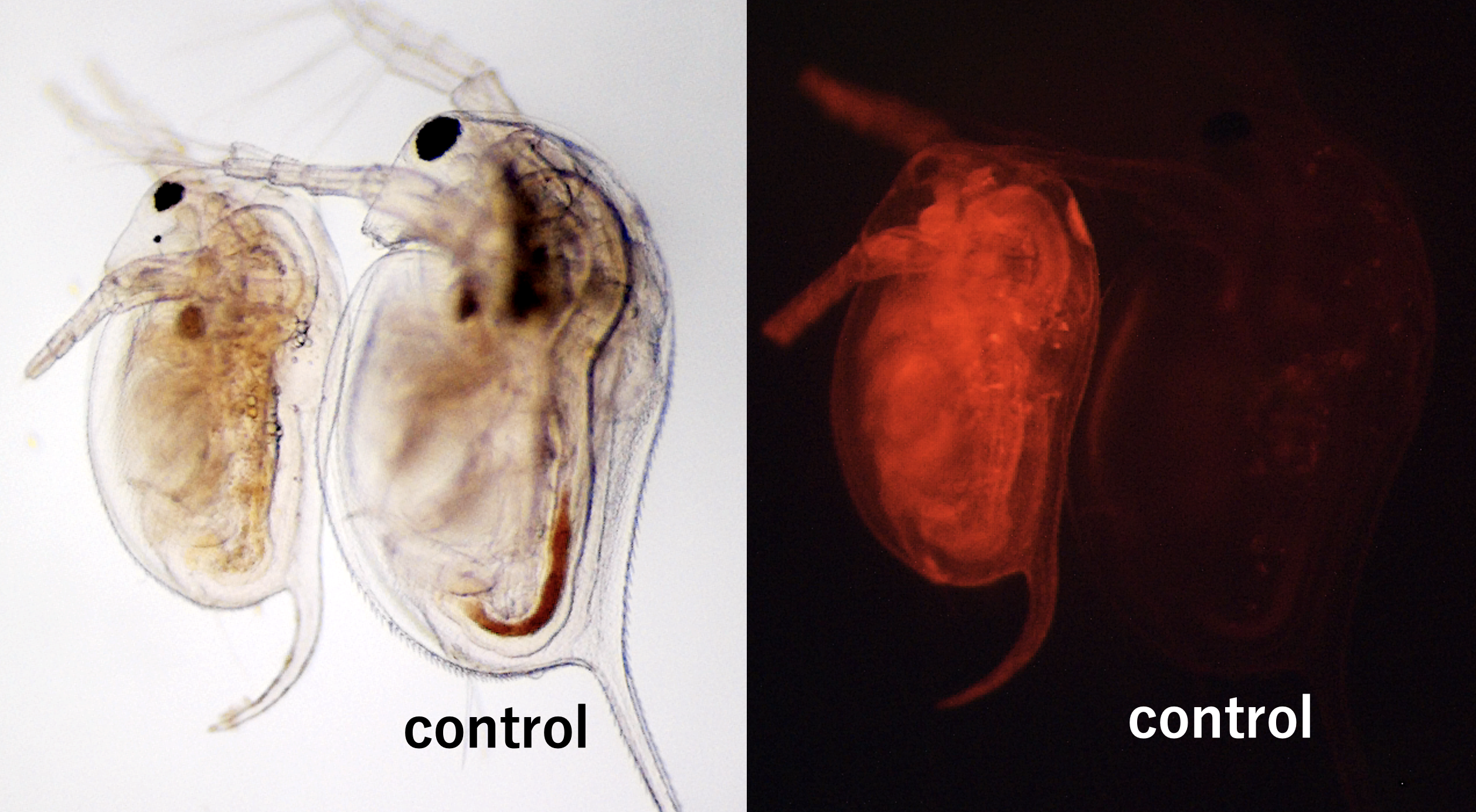
そこで polymerase を変え、T7 RNA polymerase を使って RNA を合成しました。完全長の RNA のみを合成することができ、早速インジェクション。ところが、今度は一向に光りません。。。そこで先ほどの SP6 転写産物中の short RNA の3′ 末端をシーケンスにより決定しました。すると short RNA は 522 塩基からなる EF1alpha1 3′ UTR の 130 塩基目付近での早期の転写終結により生じた RNA であることが判明しました。さらに EF1alpha1 3′ UTR 中には実は mRNA の分解や翻訳抑制に関与する AU-rich element(ARE)や Musashi binding site(MBE)が存在し、これらのエレメントが shrot RNA では除かれていたことが明らかになったのです。その後、short RNA のみのインジェクションにより DsRed2 発現が可能であることを確認、タンパク発現に最適な EF1alpha1 3′ UTRの長さを検証し、現在は EF1alpha1 3′ UTR から ARE と MBE を完全に取り除いた配列(60 塩基)を過剰発現による遺伝子機能獲得実験に使っております 。そのような不安定な EF1alpha1 mRNA の生理的意義は謎ですが(そこまで踏み込めると面白いと思っておりますが)、遺伝子発現における UTR の威力が強く心に刻まれました。
ちなみに、今改めて振り返ると、ミジンコのオス決定遺伝子 Dsx1 の発現制御機構を明らかにする上でプロモーター解析に焦点を当てるだけでなく、UTR にも性特異的な遺伝子発現に寄与するエレメントがあるはず!! と考えるきっかけとなり、本領域で研究をさせていただいております lncRNA、ダパールの発見に繋がった大事な研究結果であったと考えています。


-320x180.png)