#20の大迷走(3)
2016年に北大に異動して半年たち、最初の研究室配属で6名の学部3年生が研究室にやってきました。スタッフは僕と講師の米田さんの2名。助手時代に学生の実験指導をしたことはありましたが、各学年1名がMaxだったので、これだけの人数をいっときに抱えるのは初めての経験でてんやわんや。理研時代は学生さんがなかなか来てくれなかったので、嬉しい悲鳴ではありましたが、とかく世間は「ちょうど良い」という具合にはなかなかならないところが皮肉なものです。
ともあれ、ここで長らく塩漬けになっていた#20プロジェクトを引き継いでくれたのが、今回の論文の筆頭著者の高橋くんです。米田さんの指導のもと、#20のタンパク質産物に注目して、細胞に発現させたり、相互作用因子をとってきたりという仕事を開始しました。で、ある日、こんな論文があるんですけど、、、と言って彼が持ってきてくれた論文が、こちら。

な、なぬー??#20がもう論文になってる??なんとなんと、6330403K07Rikの名前がアブストラクトに入ってる論文が2015年に出ているではありませんか!塩漬け期間、関連研究の文献検索をupdateしてこなかったので全く気づかなかったのですが、やられた!と、心臓がバクバクしているのを必死に落ち着かせてページを繰っていくと、、、
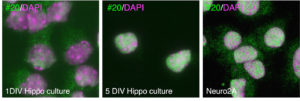
なんじゃこれ。発現パターンがぜんぜん違う。
この論文の趣旨はtanycyteという特殊なグリア細胞の特異的なマーカーが見つかりましたよ、という話です。それが6330403K07Rikであると。
えっ?#20って脳においては神経細胞ほぼ全てで発現してますけど。何か?
論文のマテメソをよくよく調べてみると、プローブを作るためにPCRで使ったプライマーの配列がゲノムの配列と1塩基だけ違います。この著者の人達、間違えて別の遺伝子をPCRで引っ掛けたんじゃないの?これまでのデータはどこをどう見てもbrain全体で発現しているし、それはAllen Brain Atlasのデータとも合っているし、彼らのin situのデータでは核内にシグナルが見えないし、原因はわからないけれどもとりあえず違うものを見ているはずでしょう、僕らは僕らで気にせず仕事を進めていけばいいんじゃないでしょうかねえ、というのが僕の意見でした。
ただ、高橋くんはこの見当外れな僕のコメントに納得せず、brainの切片を作り直してtanycyteでも発現していますよ、というデータを出してきました。tanycyteは間脳の一部にしか存在しておらず、僕らが普段使っている切片には入っていません。たしかに彼のデータを見ると、tanycyteのところにもくっきりとシグナルが見えます。この事実をどう考えるか。核内繋留されていたmRNAがtanycyteではなぜか細胞質に輸送されていて、この細胞でだけタンパク質に翻訳されている?にしても、Ma et al.論文ではtanycyte以外のシグナルはほとんどないので、話が合いません。ここで、我々はそもそも根本的に大きな勘違いをしていたという決定的なデータを、高橋くんが出してきました。我々はノックアウトマウスを持っています。そのマウスでin situをすれば、決着はつくはず。
なんだこれは。欠失変異で作ったノックアウトマウスのはずなのに、シグナルがバキバキに出てる。。。

まるで怪談です。僕らが見ていたシグナルは一体何だったのだ。さらにノザンブロットの結果が追い打ちをかけます。

#20プローブは6330403K07Rik以外に、何かよくわからないバンドをいっぱい検出しています。というより、本物のバンドよりもそちらのバンドのほうがシグナルが強いぐらい。で、これらのバンドは当然ながら、ノックアウトマウスでは消えていません。高橋くんは色々プローブの領域を変えてノザンを繰り返し、特異的なバンドのみを検出するプローブ領域を見つけました。こちらはきれいにKOでバンドが消えています。
brainの切片でも、tanycyteのシグナルを特異的に検出し、KOではきれいに消えています。

ハイブリダイゼーションの非特異的なシグナルでよくあるのは、リピート配列入ってました、という凡ミスで、僕もin situ実験を始めた頃はこれでよくハマっていたのですが、repeat maskerで検出される配列を避けていれば、基本的に非特異的なシグナルは回避できます。実際、#20プローブの配列でblastをかけても、ゲノム上の単一配列しかヒットしません。(TCTCA)nに弱いホモロジーを示す領域もありますが、その領域を含んだプローブでも特異的に6330403K07Rikを検出することも高橋くんはきれいにノザンブロットで示しています。つまり、#20プローブはblastでは予測できない配列にクロスしている、もしくは、mm10ゲノムにはアノテーションされていない未知のゲノム配列があって、そこから転写されているRNAにクロスしているといることになります。
これでこれまでの謎は全てキレイに説明できます。Neuro2Aで核内でバキバキにシグナルが見えていたのにタンパク質が抗体で検出できなかったのは、ただ単にNeuro2Aが#20プローブが検出する謎のRNAを発現していて6330403K07Rikを発現していなかっただけ。外来のmRNAを発現させたときに核内に繋留されず細胞質に運ばれていたのは、ただ単に普通のmRNAを強制発現させたものを検出していたので当たりまえだのクラッカー。6330403K07Rikは先行論文どおり、tanycyteで強く発現する、細胞タイプ特異的な小胞体局在タンパク質、ということで、そのmRNAには何ら特殊な制御がかかっているわけではない、ということが明らかになりました。
というわけで、#20プローブのシグナルに導かれて始まった研究は迷走に迷走を続け、lncRNA研究>核内繋留mRNA研究>tanycyte特異的遺伝子の研究と、もはやはじめの戦型が原型をとどめていない局面まで来てしまいました。なんで最初にノザンをしなかったのだろう。ノザンブロット大事だよ、というのが今回の最大の教訓でしょうか。
RNA研究ではなくなってしまったものの6330403K07Rikの研究が面白くない、というわけではありません。このタンパク質、C末端側に疎水性部分があり小胞体に局在しますが、細胞質部分に突き出ている部分は他のタンパク質と相同性はなく、構造を取りにくい天然変性領域になっています。ポジティブなチャージとネガティブなチャージが交互に並んでいて、いかにも非ドメイン型の相互作用をしそうな分子です。高橋くんと米田さんの実験で、免疫沈降するとなぜかミトコンドリアのATPase複合体がごっそり落ちてくることなども分かってきました。ほんと謎だらけの遺伝子です。ノックアウトマウスは外見上は大きな異常は示しませんでしたが、tanycyteは栄養状態に応じたホルモン応答や神経ペプチドの分泌に重要な役割を果たしていると考えられており、そのあたりを詳しく調べていけば、何らかの表現型が見える可能性もあるのではないかと考えています。ただ、惜しくもここで高橋くんの修士課程は時間切れ。「なつぞら」のモデルにもなった北海道が誇る酪農企業よつば乳業に就職が決まっていた彼は3月の最終週まで実験を続け、膨大なデータとともに惜しまれつつ研究室を去っていきました。論文というのはどこで区切りをつけるのかはとても難しくて、今回も、もう少しマウスの解析を粘ってからとも思ったのですが、ひとまずここでまとめることにした次第です。
しかし、#20プローブで見えるあのへんなシグナル、一体何だったのでしょう。スメアなシグナルではないので、なにか特異的な核局在RNAを引っ掛けているはずです。それがなにか、正体を明かしたい気もしますが、そういう思いつきの研究はドツボにはまりますよという教訓が得らればかりなので、いまはそっと頭の片隅に置いておくだけにしようかと思っています。でもきっと気になってまたちょこちょこ実験始めてしまうんだろうなあとは思いつつ。。。(完)
投稿者プロフィール

最新の投稿
 ノンドメインブログ2025.06.27来し方行末~パート1
ノンドメインブログ2025.06.27来し方行末~パート1 ノンドメインブログ2025.02.11平和な日々
ノンドメインブログ2025.02.11平和な日々 ノンドメインブログ2024.11.25MBSJ2024のシンポジウムのすごいゲスト
ノンドメインブログ2024.11.25MBSJ2024のシンポジウムのすごいゲスト ノンドメインブログ2024.09.17奇遇癖
ノンドメインブログ2024.09.17奇遇癖